|
|
 |
細胞内のシグナル伝達機構を分子レベルで明らかにする試み |
 |
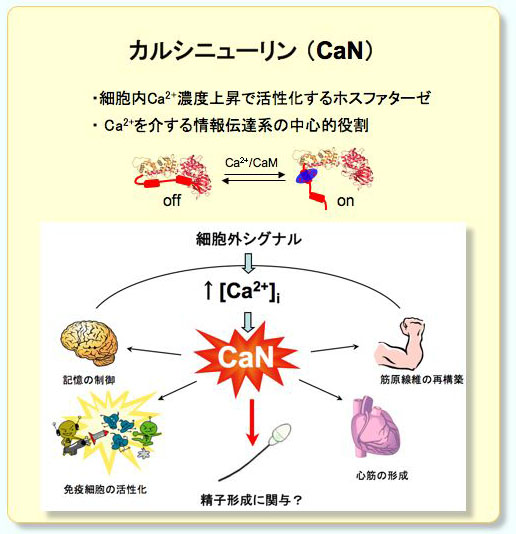
Ca2+を介した細胞内シグナル伝達とカルシニューリン |
 |
カルシニューリンは、Ca2+/カルモジュリンにより酵素活性が直接調節されるセリン/スレオニンホスファターゼ(タンパク質脱リン酸化酵素)です。この酵素は遺伝子発現制御からタンパク質の機能調節まで、Ca2+シグナルを介した種々の細胞内シグナル伝達経路で重要な働きを担っており、免疫応答、神経伝達効率の調節やストレス応答、筋細胞形成など、生体の様々な機能に関わっています。カルシニューリンは、酵母からヒトにいたる真核生物に普遍的に存在しており、カルシニューリンの関与する細胞内シグナル伝達経路は生物に共通の機構であることがわかってきています。
|
 |
カルシニューリンとカルシニューリン調節タンパク質 |
 |
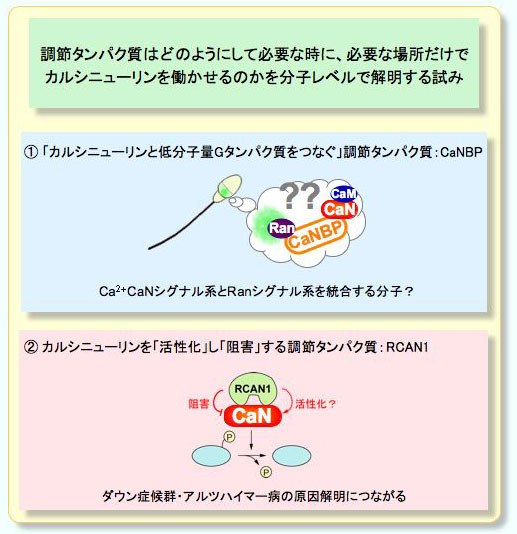
セリン/スレオニンホスファターゼは、およそ400種類存在するセリン/スレオニンキナーゼ(タンパク質リン酸化酵素)に対して25種類程と非常に少ないのですが、調節タンパク質と複合体を形成することで多様性を作り出していることが近年明らかになってきました。つまり、一つの酵素が複数の調節タンパク質によって、活性や細胞内局在、組織特異性を様々に制御されているのです。生物有機化学研究室では、カルシニューリンの調節タンパク質に注目して、調節タンパク質がどのようにして必要な時に必要な場所だけでカルシニューリンを働かせるのか、そのメカニズムを明らかにようとしています。
|
 |
 |
 |
 |
①「カルシニューリンと低分子量Gタンパク質をつなぐ」調節タンパク質 |
 |
カルシニューリンには、精巣特異的アイソフォームが存在すること、精子形成時に発現量や局在が変化することなどから、発見当初より精巣で重要な役割をもつことが推測されていますが、具体的な機能は一切明らかになっていません。私たちは、精巣におけるCaNの機能解明の糸口となると考えられるCaN結合タンパク質(CaNBP:CaNBP75およびRanBP3)を見出し、このタンパク質が、核-細胞質間分子輸送の調節タンパク質として知られる低分子量Gタンパク質Ranとも結合することを発見しました。Ranは、近年、紡錘体形成や分裂後の核膜の再構成など、体細胞分裂の多くのプロセスでスイッチ分子として関わることが明らかになってきています。私たちは、CaNBPがCaNを介したCa2+/カルモジュリンシグナル伝達系と、GTP/GDP-Ranシグナル系を統合するリンカーとして、重要な働きを持つのではないだろうか、という仮定のもと、CaN-CaNBPs-Ran相互作用機構、および(精巣の)CaNの機能解明に取り組んでいます。
|
 |
②カルシニューリンを「活性化」し「阻害」する調節タンパク質 |
 |
カルシニューリン調節タンパク質の一つであるRCAN1は、カルシニューリン経路を持つ多くの真核生物において共通に存在することがわかっています。このタンパク質は、標的のカルシニューリンを『活性化』し、かつ、『阻害する』という相反する機能を持つことが示されていますが、その調節機構についてはまだ解明されていません。RCAN1が関与するとされる疾病の中でも特に、RCAN1によるカルシニューリンの阻害がダウン症候群の原因のひとつであると考えられており、具体的な機構の解明が待たれています。
生物有機化学研究室では、様々なRCAN1変異体を用いて、機能解析と構造解析の両面から、RCAN1による正・負のカルシニューリン調節機構を解明しようとしています。
|
 |
 |
 |
 |
論文 etc. |
 |
【原著論文】 |
 |
| Hino H, Arimoto K, Yazawa M, Murakami Y, Nakatomi A. (2012) |
| Ran and Calcineurin Can Participate Collaboratively in the Regulation of Spermatogenesis in Scallop.
|
| Mar Biotechnol[ Epub ahead of print]
|
| (PMID:22246556) |
| Kitagawa C, Nakatomi A, Hwang D, Murakami Y, Ohki S. (2011) |
| Roles of the C- Terminal Residues of Calmodulin in Structure and Function.
|
| Biophysics 7, 35- 49
|
| (doi:10.2142/biophysics.7.35) |
| Isozumi N, Iida Y, Nakatomi A, Nemoto N, Yazawa M, Ohki S. (2011) |
| Conformation of the calmodulin-binding domain of metabotropic glutamate receptor subtype 7 and its interaction with calmodulin.
|
| J Biochem. 149(4), 463-74
|
| (PMID:22019463) |
| Graidist P, Yazawa M, Tonganunt M, Nakatomi A, Lin CC, Chang JY, Phongdara A, Fujise K. |
| Fortilin binds Ca2+ and blocks Ca2+-dependent apoptosis in vivo.
|
| Biochem J. 408(2), 181-91 (2007)
|
| (PMID:17705784) |
| Kurita H., Nakatomi A., Shimahara H., Yazawa M., Ohki SY. |
| Al(3+) interaction sites of calmodulin and the Al(3+) effect on target binding of calmodulin. |
| Biochem Biophys Res Commun. 333(4), 1060-1065 (2005) |
| (PMID:15964544) |
| Moriya M., Ochiai M., Yuasa HJ., Suzuki N., and Yazawa M., (2004) |
| Identification of Ca2+-Dependent Calmodulin-Binding Protein in Rat Spermatogenic Cells as Complexes of the Heat-Shock Proteins. |
| Mol. Reprod. Dev. 69, 316-324 (2004) |
| (PMID:15349844) |
| Nakatomi A. and Yazawa M. |
| Identification and Characterization of a Novel Calcineurin-Binding Protein in Scallop Testis. |
| J. Biochem. (Tokyo) 133(2), 159-164 (2003) |
| (PMID:12761177) |
| Yokouchi, T., Nogami, H., Izumi, Y., Yoshino, H., Nakashima, K., and Yazawa, M. |
| Solution X-ray scattering data show structural differences among chimeras of yeast and chicken calmodulin: implications for structure and function. |
| Biochemistry 42(7), 2195-2201 (2003) |
| (PMID:12590609) |
| Ishida H, Nakashima K, Kumaki Y, Nakata M, Hikichi K, and Yazawa M. |
| The solution structure of apocalmodulin from Saccharomyces cerevisiae implies a mechanism for its unique Ca2+ binding property. |
| Biochemistry 41(52), 15536-15542 (2002) |
| (PMID:12501182) |
| Yuasa HJ, Nakatomi A, Suzuki T, and Yazawa M. |
| Genomic structure of the sponge, Halichondria okadai calcyphosine gene. |
| Gene 298(1), 21-7 (2002) |
| (PMID:12406572) |
| Ishida H, Nakashima K, Kumaki Y, Nakata M, Hikichi K, and Yazawa M. |
| 1H, 15N and 13C resonance assignments of yeast Saccharomyces cerevisiae calmodulin in the Ca2+-free state. |
| J. Biomol. NMR 23(4), 323-324 (2002) |
| (PMID:12398353) |
| Yazawa M. |
| Quantitative analysis of Ca(2+)-binding by flow dialysis. |
| Methods Mol. Biol. 173, 3-14 (2002) |
| (PMID:12398353) |
| Yuasa HJ, Suzuki T, Yazawa M. |
| Structural organization of lower marine nonvertebrate calmodulin genes. |
| Gene 279(2), 205-212 (2001) |
| (PMID:11733145) |
| Hayashi Y, Senba S, Yazawa M, Brautigan DL, Eto M. |
| Defining the structural determinants and a potential mechanism for inhibition of myosin phosphatase by the protein kinase C-potentiated inhibitor protein of 17 kDa. |
| J. Biol. Chem. 276(43), 39858-39863 (2001) |
| (PMID:11517233) |
| Ishida H, Takahashi K, Nakashima K, Kumaki Y, Nakata M, Hikichi K, and Yazawa M. |
| Solution structures of the N-terminal domain of yeast calmodulin: Ca2+-dependent conformational change and its functional implication |
| Biochemistry 39, 13660-13668 (2000) |
| (PMID:11076504) |
| Uryu M, Nakatomi A, Watanabe M, Hatsuse R, Yazawa M. |
| Molecular cloning of cDNA encoding two subunits of calcineurin from scallop testis: demonstration of stage-specific expression during maturation of the testis |
| J. Biochem. 127, 739-746 (2000) |
| (PMID:10788781) |
|
|

